LDA เป็นเทคนิคในการจัดกลุ่ม (classification) โดยการสร้างโมเดลจาก training set เพื่อใช้ทดสอบการจัดกลุ่มในตัวอย่างในอนาคต ในตัวอย่างก่อนหน้านี้ซึ่งเราต้องการจัดกลุ่มตัวอย่างออกเป็น 3 กลุ่ม เราจะแบ่งข้อมูลตัวอย่างจำนวน 300 ตัวอย่างเป็น Training 150 ตัวอย่าง และอีก 150 ตัวอย่างไว้ทดสอบ
> coffee<-csv2matrix()
> coffee.df<-as.data.frame(coffee)
> groups<-rep(c("good","unpeel","black"),each=100)
สร้างตัวแปร เลขคู่ เลขคี่เพื่อแบ่งตัวอย่าง (อาจจะไม่ใช่วิธีการที่ดีในการแบ่ง ไว้เราค่อยหาวิธีการแบ่งตัวอย่างที่ดีกว่านี้)
> even<-seq(2,300,2)
> odd<-seq(1,300,2)
แบ่งข้อมูลเป็น 2 ชุด
> coffee.trn <-coffee.df[odd,]
> coffee.test <-coffee.df[even,]
ใช้ฟังก์ชั่น lda ในแพกเกจ MASS
> install.packages("MASS")
> library(MASS)
> coffee.lda <- lda(coffee.trn,grouping =groups[odd],prior = rep(1,3)/3)
Warning message:
In lda.default(x, grouping, ...) : variables are collinear
เกิดความข้อความเตือนเนื่องจากตัวแปร x ใน NIR มีลักษณะ collinear คือมีความสัมพันธ์กันเอง เราจะแก้ปัญหานี้ในภายหลัง
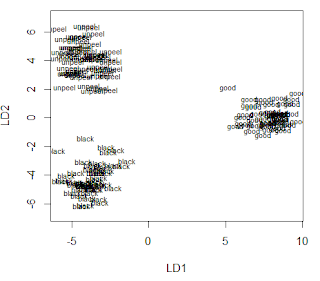
> plot(coffee.lda)
จากการพล็อตข้อมูลของ Training set จะเห็นว่าสามารถแยกกลุ่มออกได้ชัดเจนมาก
ทำนายการจัดกลุ่มตัวอย่างอีก 150 ตัวอย่างด้วยฟังก์ชั่น predict
> coffee.predict <-predict(coffee.lda,new=coffee.test)
แสดงผลการทำนายกลุ่ม
> table(groups[even],coffee.predict$class)
black good unpeel
black 50 0 0
good 0 50 0
unpeel 0 0 50
จะพบว่าโมเดลที่สร้างขึ้นสามารถจัดกลุ่มตัวอย่างใน test set ได้ถูกต้อง 100 %
เนื่องจากตัวแปรของ NIR มีจำนวนมากและมักจะมีลักษณะสัมพันธ์กัน เราสามารถใช้ตัวแปรใหม่จาก PCA นั่นคือ PC มาใช้ในการทำ LDA ได้
> coffee.pca.trn <-prcomp(coffee.trn)
> summary(coffee.pca.trn)
predict ค่า PC ใน test set
> coffee.pca.test<-predict(coffee.pca.trn,new=coffee.test)
จาก summary จะพบว่า PC1-10 อธิบายความแปรปรวนได้เกิน 99% ดังนั้นเราจะใช้เพียง PC1-10 เป็นตัวแปรสำหรบ LDA
> coffee.pca.lda <- lda(coffee.pca.trn$x[,1:10],grouping =groups[odd],prior = rep(1,3)/3)
> plot(coffee.pca.lda)
> coffee.pca.predict <-predict(coffee.pca.lda,new=coffee.pca.test[,1:10])
> table(groups[even],coffee.pca.predict$class)
black good unpeel
black 50 0 0
good 0 50 0
unpeel 0 0 50
แสดงบทความที่มีป้ายกำกับ PCA แสดงบทความทั้งหมด
แสดงบทความที่มีป้ายกำกับ PCA แสดงบทความทั้งหมด
วันเสาร์ที่ 12 ธันวาคม พ.ศ. 2558
วันพุธที่ 9 ธันวาคม พ.ศ. 2558
วิเคราะห์ PCA ด้วย R
เราสามารถวิเคราะห์ PCA (Principal Component Analysis) บน R ได้หลายวิธี แต่วิธีที่นิยมคือการใช้ ฟังก์ชั่น prcomp
ในตัวอย่างนี้ผมจะลองวิเคราะห์ PCA เพื่อแบ่งกลุ่ม NIR spectra ของตัวอย่าง 3 กลุ่ม กลุ่มละ 100 ตัวอย่าง ในแต่เส้น NIR spectrum นั้นได้มากจากการวัดการดูดกลืนแสงในช่วง12500 - 4000 cm-1 จึงมีข้อมูลจำนวน 558 ค่า ข้อมูลซึ่งเดิมมาจากโปรแกรม OPUS ถูกแปลงเป็น csv ตามโพสต์ก่อนหน้านี้ จากนั้นแปลง csv เป็น matrix อีกที
จาก matrix เราจะแปลงข้อมูลเป็น data.frame เพื่อให้ง่ายต่อการวิเคราะห์
> source("D:\\csv2matrix.R")
> data<-csv2matrix()
> data.df<-as.data.frame(data)
สร้างข้อมูลจัดกลุ่มตัวอย่าง 3 กลุ่ม คือ good, black, unpeel
> groups<-rep(c("good","black","unpeel"),each=100)
วิเคราะห์ PCA ด้วยฟังก์ชั่น prcomp()
> data.pca<-prcomp(data.df)
แสดง PC ต่างๆ
> summary(data.pca)
จากนั้นเราจะแสดง biplot ด้วย ggbiplot ซึ่งจะต้องติดตั้งจาก github
> library(devtools)
> install_github("vqv/ggbiplot")
> library(ggbiplot)
> ggbiplot(data.pca,obs.scale=0,groups=groups,ellipse=TRUE,var.axes=FALSE)
ในตัวอย่างนี้ผมจะลองวิเคราะห์ PCA เพื่อแบ่งกลุ่ม NIR spectra ของตัวอย่าง 3 กลุ่ม กลุ่มละ 100 ตัวอย่าง ในแต่เส้น NIR spectrum นั้นได้มากจากการวัดการดูดกลืนแสงในช่วง12500 - 4000 cm-1 จึงมีข้อมูลจำนวน 558 ค่า ข้อมูลซึ่งเดิมมาจากโปรแกรม OPUS ถูกแปลงเป็น csv ตามโพสต์ก่อนหน้านี้ จากนั้นแปลง csv เป็น matrix อีกที
จาก matrix เราจะแปลงข้อมูลเป็น data.frame เพื่อให้ง่ายต่อการวิเคราะห์
> source("D:\\csv2matrix.R")
> data<-csv2matrix()
> data.df<-as.data.frame(data)
สร้างข้อมูลจัดกลุ่มตัวอย่าง 3 กลุ่ม คือ good, black, unpeel
> groups<-rep(c("good","black","unpeel"),each=100)
วิเคราะห์ PCA ด้วยฟังก์ชั่น prcomp()
> data.pca<-prcomp(data.df)
แสดง PC ต่างๆ
> summary(data.pca)
จากนั้นเราจะแสดง biplot ด้วย ggbiplot ซึ่งจะต้องติดตั้งจาก github
> library(devtools)
> install_github("vqv/ggbiplot")
> library(ggbiplot)
> ggbiplot(data.pca,obs.scale=0,groups=groups,ellipse=TRUE,var.axes=FALSE)
สมัครสมาชิก:
บทความ (Atom)